2022年5月30日,黄俊实验室在Nucleic Acids Research杂志上在线发表了题为“RNF4 controls the extent of replication fork reversal to preserve genome stability”的研究论文,揭示了细胞调控复制叉翻转及维持基因组稳定性的分子机制。
DNA的精确复制对维持基因组稳定性以及保证遗传信息在世代间的准确传递至关重要。复制叉是DNA复制的基本单位,DNA在复制过程中时常受到各种内源性或者外源性的复制压力影响而导致复制叉停滞,复制叉翻转(fork reversal)是高等真核生物在进化上保守的一种复制压力应答机制。黄俊实验室在2021年的Molecular Cell研究中首次提出了复制叉翻转的两步级联模型:第一步,在遭遇复制压力时,复制叉发生停滞,由转位酶SMARCAL1、HLTF、ZRANB3以及重组酶RAD51等蛋白催化复制叉的初始翻转,同时在复制叉后方新合成的DNA双链上产生拓扑张力;第二步,拓扑异构酶TOP2A释放拓扑张力,并通过ZATT对其的SUMO化修饰招募转位酶PICH促进复制叉的深度翻转。
深度翻转的复制叉能够帮助细胞应对复制压力,保护基因组的稳定性,但如果TOP2A-ZATT-PICH不受调控地促进深度翻转的复制叉持续翻转,则会导致复制叉过度翻转,复制压力移除后复制叉无法正常重启,同样会威胁基因组的稳定性。目前细胞调控复制叉翻转程度的分子机制并不清楚。此研究在该实验室2021年Molecular Cell工作的基础上发现,在复制压力下,SUMO靶向的泛素E3连接酶RNF4通过SIM结构结合SUMO化饰的TOP2A,随后对其进行多聚泛素化修饰,促进其降解。RNF4抑制TOP2A-ZATT-PICH在深度翻转的复制叉上过度累积从而抑制了复制叉的过度翻转。该研究完善了复制叉翻转及调控的分子机制,揭示了高等真核生物在复制压力下保证DNA正常复制、遗传信息准确传递的机理。
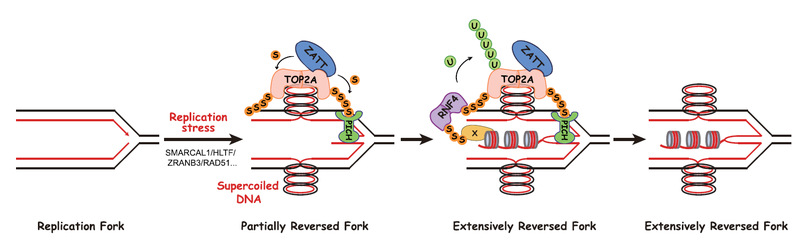
图:RNF4调控复制叉翻转的分子机制
黄俊实验室的博士研究生丁林丽、罗艺、阳玉兰,已经毕业的博士研究生陈旭以及中山大学第八附属医院的博士后田甜为论文共同第一作者,黄俊教授为通讯作者。该研究受到国家科技部重点研发计划、国家自然科学基金、霍英东青年教师基金等项目的资助。
原文链接:https://doi.org/10.1093/nar/gkac447

